02. Dezember 2024
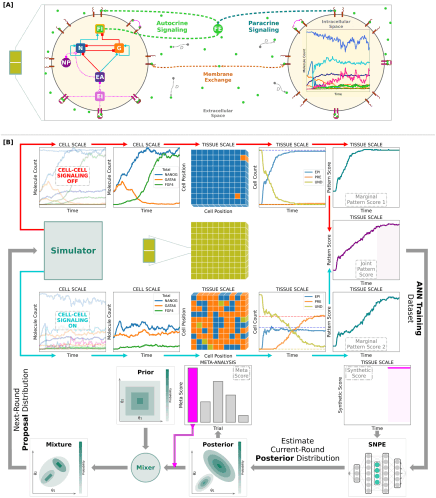
In Kollaboration mit Maciej Majka und Marcin Zagórski (Uni Krakau), Nils Becker (DKFZ Heidelberg) und Pieter Rein ten Wolde (AMOLF, Amsterdam) publizieren wir den Artikel "Stable developmental patterns of gene expression without morphogen gradients", ebenfalls in PLoS Computational Biology.